Proteine üben nicht nur Funktionen aus, die für das Überleben von Zellen entscheidend sind, sondern beeinflussen auch die Entstehung und den Verlauf von Krankheiten. Um ihre Rolle bei Gesundheit und Krankheit zu verstehen, untersuchen Forscher die dreidimensionale atomare Struktur von Proteinen sowohl mit experimentellen als auch mit rechnerischen Methoden.
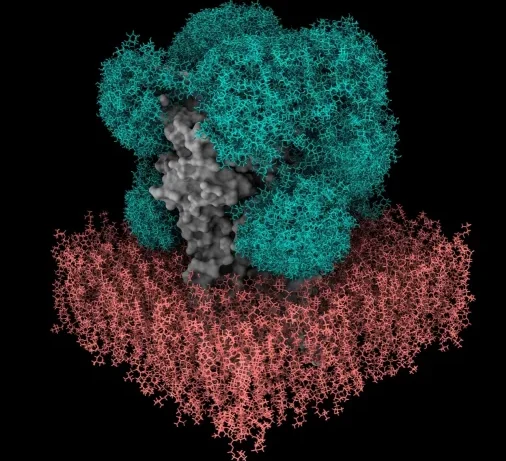
Über 75 Prozent der Proteine an der Oberfläche unserer Zellen sind mit Glykanen bedeckt. Diese zuckerähnlichen Moleküle bilden sehr dynamische Schutzschilde um die Proteine. Aufgrund der Mobilität und Variabilität der Zucker ist es jedoch schwierig zu bestimmen, wie sich diese Schutzschilde verhalten oder wie sie die Bindung von Arzneimittelmolekülen beeinflussen.

Mateusz Sikora, der Projektleiter und Leiter des Dioscuri Centre for Modelling of Posttranslational Modifications, und sein Team in Krakau sowie Partner am Max-Planck-Institut für Biophysik in Frankfurt am Main haben diese Herausforderung mit Hilfe von Computern angegangen und dabei mit Wissenschaftlern des Inserm in Paris, der Academia Sinica in Tapei und der Universität Bremen zusammengearbeitet.
Ihr leistungsstarker neuer Algorithmus GlycoSHIELD ermöglicht eine schnelle, aber realistische Modellierung der Zuckerketten auf Proteinoberflächen. GlycoSHIELD reduziert die Rechenzeit und damit den Stromverbrauch im Vergleich zu konventionellen Simulationswerkzeugen um mehrere Größenordnungen und ebnet damit den Weg zum Green Computing.
Von Tausenden von Stunden auf wenige Minuten
Schützende Glykanschilde haben einen großen Einfluss darauf, wie Proteine mit anderen Molekülen, z. B. therapeutischen Medikamenten, interagieren. So verbirgt beispielsweise die Zuckerschicht auf dem Spike-Protein des Coronavirus das Virus vor dem Immunsystem, indem sie die Erkennung des Virus durch natürliche oder impfstoffinduzierte Antikörper erschwert. Die Zuckerschilde spielen daher eine wichtige Rolle bei der Entwicklung von Medikamenten und Impfstoffen. Die pharmazeutische Forschung könnte von einer routinemäßigen Vorhersage ihrer Morphologie und Dynamik profitieren. Bislang war die Vorhersage der Struktur von Zuckerschichten mit Hilfe von Computersimulationen jedoch nur mit Expertenwissen auf speziellen Supercomputern möglich. In vielen Fällen waren dafür Tausende oder gar Millionen von Rechenstunden erforderlich.
Mit GlycoSHIELD bietet Sikoras Team eine schnelle, umweltfreundliche Open-Source-Alternative. „Unser Ansatz reduziert die Ressourcen, die Rechenzeit und das benötigte technische Fachwissen“, sagt Sikora. „Jeder kann nun die Anordnung und Dynamik von Zuckermolekülen auf Proteinen innerhalb von Minuten auf seinem PC berechnen, ohne dass er Expertenwissen und Hochleistungsrechner benötigt.
Außerdem ist diese neue Art der Berechnung sehr energieeffizient.“ Die Software kann nicht nur in der Forschung eingesetzt werden, sondern könnte auch bei der Entwicklung von Medikamenten oder Impfstoffen hilfreich sein, zum Beispiel bei der Immuntherapie gegen Krebs.
Ein Puzzlespiel aus Zucker
Wie ist es dem Team gelungen, eine so hohe Effizienzsteigerung zu erreichen? Die Autoren erstellten und analysierten eine Bibliothek mit Tausenden von wahrscheinlichen 3D-Positionen der häufigsten Formen von Zuckerketten auf Proteinen, die in Menschen und Mikroorganismen vorkommen. Anhand langer Simulationen und Experimente fanden sie heraus, dass es für eine zuverlässige Vorhersage von Glykanabschirmungen ausreicht, dass die angehängten Zucker nicht mit Membranen oder Teilen des Proteins kollidieren.
Der Algorithmus basiert auf diesen Erkenntnissen. „GlyoSHIELD-Benutzer müssen nur das Protein und die Stellen, an denen die Zucker gebunden sind, angeben. Unsere Software puzzelt sie dann auf der Proteinoberfläche in der wahrscheinlichsten Anordnung zusammen“, erklärt Sikora. „Wir konnten die Zuckerschilde des Spike-Proteins genau nachbilden: Sie sehen genauso aus, wie wir sie in den Experimenten sehen!“ Mit GlycoSHIELD ist es nun möglich, sowohl neue als auch bestehende Proteinstrukturen mit Zuckerinformationen zu ergänzen. Die Wissenschaftler nutzten GlycoSHIELD auch, um das Muster der Zucker auf dem GABAA-Rezeptor, einem wichtigen Ziel für Beruhigungs- und Narkosemittel, aufzudecken.